12月8日,Nature 旗下的国际学术期刊Cell Research (2014年影响因子12.413
)在线发表了国家重点实验室杨东雷教授为第一作者的题为"Dicer-independent RNA-directed DNA methylation in Arabidopsis”的研究论文,本论文与中科院上海植物逆境生物学研究中心,美国科学院院士-朱健康合作完成。
DNA 甲基化是调控基因和转座子表达的重要表观遗传标记,在模式植物-拟南芥中,DNA甲基化由两个并列的分子途径负责从头起始,分别为RNA-Directed DNA Methylation (RdDM) 途径和DDM1-CMT2途径。现在普遍认为RdDM途径由24-nt siRNA来介导,因此人们常常将24-nt siRNA的积累量跟DNA甲基化水平联系起来。 虽然大家早就注意到负责切割产生24-nt siRNA 的Dicer like 3 (DCL3) 的突变体的甲基化下降比预期的要明显的低,但是人们推测这是由DCL3的同源基因功能冗余造成的。在本研究中,杨东雷和张贵平等同事将拟南芥基因组中的四个DCL基因全部敲除,与预期相同的是四突变的中的24-nt siRNA几乎全部消失,但是出乎意料的是它的甲基化水平并没有下降到 RdDM其他突变体(pol iv, pol v, rdr,drm2)相同低的水平,在很多位点上甚至达到了跟野生型对照的相同的甲基化水平,这就推翻了被广泛认可的观念即24-nt siRNA对于RdDM是必需的。
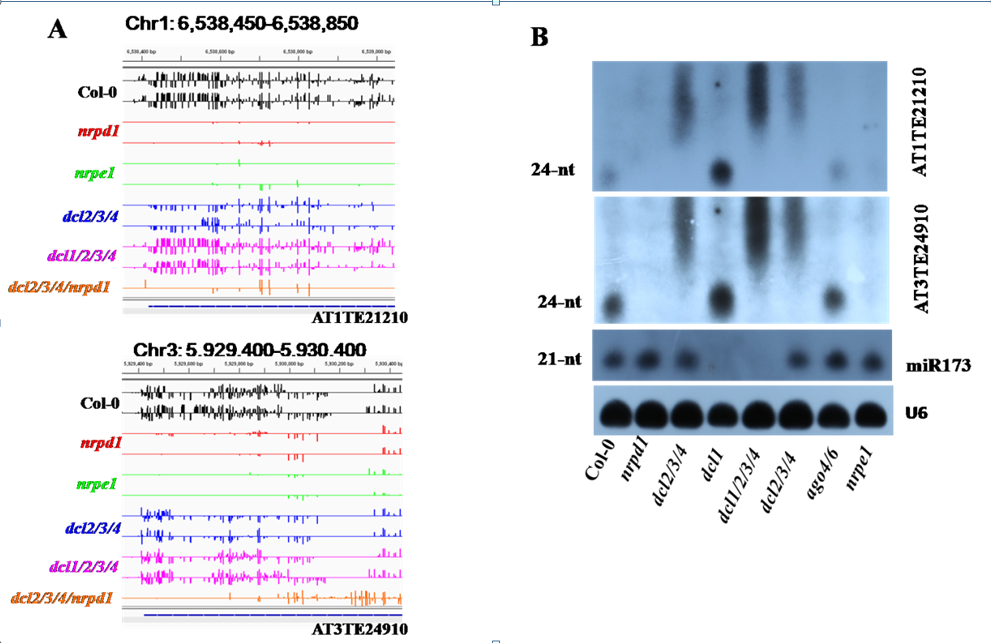
图 两个ID位点的甲基化水平和P4 RNA的积累
他们进一步根据dcl1/2/3/4 四突变的全基因组的甲基化信息,把拟南芥基因组的RdDM 位点分为三类:FD (fully dependent on DCLs)位点、PD (partially dependent on DCLs),ID (DCLs-independent)位点。其中完全依赖DCLs的FD位点中dcl1/2/3/4的甲基化水平与Pol IV, Pol V等突变体的水平相似,提示24-nt siRNA对于这些位点的DNA甲基化非常重要,该位点仅仅占到全部RdDM位点的15.5%。而完全不依赖DCLs的ID位点中dcl1/2/3/4的甲基化水平与野生型对照的水平相比并没有下降,表明24-nt siRNA对于DNA甲基化几乎没有贡献,但该位点却占到全基因组RdDM位点的27.4% . 而其他大约一半的RdDM位点的DNA甲基化是部分不依赖于DCLs,表明24-nt siRNA 也不是完全必需的。
通过小RNA测序他们首次发现,虽然在dcl2/3/4三突变和四突变中24-nt siRNA几乎全部消失,但是一类25-35 nt 的RNA却得到了大幅得累积,由于这些RNA的累积依赖于Pol IV,所以他们将这些新发现的RNA命名为 P4 RNA. 他们进一步详细得分析发现,在ID、PD位点位点上dcl1/2/3/4突变体比dcl2/3/4 有更多P4 RNA的累积,同时dcl1/2/3/4突变体中的ID和PD位点中DNA甲基化水平明显比dcl2/3/4突变体中相应的位点高,提示P4 RNA的量跟DNA 甲基化程度之间的相关性。
他们结合已经发布的拟南芥组蛋白修饰的全基因组数据,分析发现另外一项有意思的现象即FD位点主要位于富含活跃表观遗传标记(H3K4me2,H3K4me3,H3K9ac)即转录活跃的常染色质区,而ID, PD位点主要位于富含抑制表观遗传标记(H3K9me2,H3K27me1)的转录不活跃的异染色质区域。表明在RdDM途径中,24-nt siRNA和他们的前体P4 RNA 哪个更重要可能取决所在位点组蛋白修饰等所决定的周边染色质的环境。
这项研究的发表将改变人们对于RdDM的分子机理的理解。 |